Page 102 - 《广西植物》2020年第6期
P. 102
8 4 8 广 西 植 物 40 卷
在线软件进行分析ꎮ 此外ꎬ核苷酸与氨基端序列
的同源 性 用 DNAMAN 软 件 进 行 比 对 分 析ꎬ 并 用 2 结果与分析
Clustal X 和 MEGA 构建系统进化树ꎮ
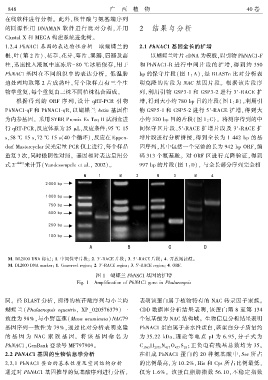
1.2.4 PhNAC1 基因的表达特性分析 取蝴蝶兰的 2.1 PhNAC1 基因全长的扩增
根、叶(第 2 片)、花葶、花芽、萼片、翼瓣、唇瓣及蕊 以蝴蝶兰叶片 cDNA 为模板ꎬ以引物 PhNAC1 ̄F
柱ꎬ迅速投入液氮中速冻后-80 ℃ 冰箱保存ꎬ用于 和 PhNAC1 ̄R 进行中间片段的扩增ꎬ得到约 350
PhNAC1 基因在不同组织中的表达分析ꎮ 低温胁 bp 的保守片段( 图 1:A)ꎬ经 BLASTn 比对分析表
迫处理均取第 2 片成熟叶ꎬ每个取样点有三个生 明克隆的片段为 NAC 基因片段ꎮ 根据该片段序
物学重复ꎬ每个重复由三株不同植株混合而成ꎮ 列ꎬ利用引物 GSP3 ̄1 和 GSP3 ̄2 进行 3′ ̄RACE 扩
根据 得 到 的 ORF 序 列ꎬ 设 计 qRT ̄PCR 引 物 增ꎬ得到大小约 780 bp 目的片段(图 1:B)ꎬ利用引
PhNAC1 ̄qF 和 PhNAC1 ̄qRꎬ以蝴蝶兰 Actin 基因作 物 GSP5 ̄1 和 GSP5 ̄2 进行 5′ ̄RACE 扩增ꎬ得到大
为内参基因ꎮ 采用 SYBR Premix Ex Taq II 试剂盒进 小约 320 bp 目的片段(图 1:C)ꎮ 将测序得到的中
行 qRT ̄PCRꎬ反应体系为 25 μLꎬ反应条件:95 ℃ 15 间保守区片段、5′ ̄RACE 扩增片段及 3′ ̄RACE 扩
sꎬ58 ℃ 15 sꎬ72 ℃ 15 s(40 个循环)ꎬ反应在 Eppen ̄ 增片段进行分析拼接ꎬ得到全长为 1 442 bp 的基
dorf Mastercycler 荧光定量 PCR 仪上进行ꎬ每个样品 因序列ꎬ其中包括一个完整的长为 942 bp ORFꎬ编
重复 3 次ꎬ同时做阴性对照ꎮ 基因相对表达量用公 码 313 个氨基酸ꎮ 对 ORF 区进行克隆验证ꎬ得到
-ΔΔCt 997 bp 的片段(图 1:D)ꎬ 与全长部分序列完全相
式 2 来计算(Vandesompele et al.ꎬ 2002)ꎮ
M. DL2000 DNA 标记ꎻ 1. 中间保守片段ꎻ 2. 3′ ̄RACE 片段ꎻ 3. 5′ ̄RACE 片段ꎻ 4. 开放阅读框ꎮ
M. DL2000 DNA markerꎻ 1. Conserved regionꎻ 2. 3′ ̄RACE regionꎻ 3. 5′ ̄RACE regionꎻ 4. ORF.
图 1 蝴蝶兰 PhNAC1 基因的扩增
Fig. 1 Amplification of PhNAC1 gene in Phalaenopsis
同ꎮ 经 BLAST 分析ꎬ 所得的核苷酸序列与小兰屿 表明该蛋白属于植物特有的 NAC 转录因子家族ꎮ
蝴蝶兰( Phalaenopsis equestrisꎬ XP _020576379) 一 CDD 数据库分析结果表明ꎬ该蛋白第 8 至第 134
致性为 98%ꎬ与小野芭蕉( Musa acuminata) NAC79 个氨基酸为 NAC 结构域ꎮ 生物信息分析结果表明
基因序列一致性为 79%ꎬ通过比对分析表明克隆 PhNAC1 蛋白属于亲水性蛋白ꎬ该蛋白分子质量约
的 基 因 为 NAC 家 族 基 因ꎮ 将 该 基 因 命 名 为 为 35.22 kDa、理 论 等 电 点 pI 为 6. 95ꎬ分 子 式 为
PhNAC1ꎬGenBank 登录号 MF797909ꎮ C H N O S ꎻ正负电荷残基总数均为 35ꎮ
1563 2375 417 477 18
2.2 PhNAC1 基因的生物信息学分析 在组成 PhNAC1 蛋白的 20 种氨基酸中ꎬSer 所占
2.2.1 PhNAC1 蛋白的基本性质及空间结构分析 的比例最高ꎬ为 10.2%ꎬHis 和 Cys 所占比例最低ꎬ
通过对 PhNAC1 基因推导的氨基酸序列进行分析ꎬ 仅为 1.6%ꎮ 该蛋白脂肪指数 56.10ꎬ不稳定指数