Page 143 - 《广西植物》2023年第10期
P. 143
10 期 施晓静等: 基于全叶绿体基因组分析的栽培黄草乌基源研究 1 8 9 7
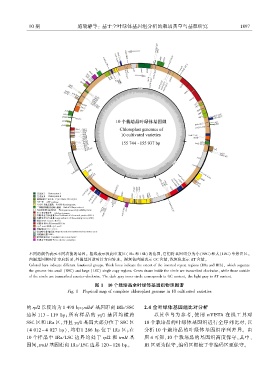
不同的颜色表示不同功能的基因ꎮ 粗线表示反向重复区(IRa 和 IRb)的范围ꎬ它们将基因组分为小(SSC)和大(LSC)单拷贝区ꎮ
内圈基因顺时针方向转录ꎬ外圈基因逆时针方向转录ꎮ 深灰色内圈表示 GC 含量ꎬ浅灰色表示 AT 含量ꎮ
Colored bars indicate different functional groups. Thick lines indicate the extent of the inverted repeat regions (IRa and IRb)ꎬ which separate
the genome into small (SSC) and large (LSC) single copy regions. Genes drawn inside the circle are transcribed clockwiseꎬ while those outside
of the circle are transcribed counter ̄clockwise. The dark gray inner circle corresponds to GC contentꎬ the light gray to AT content.
图 1 10 个栽培品全叶绿体基因组物理图谱
Fig. 1 Physical map of complete chloroplast genome in 10 cultivated varieties
的 rpl2 长度均为 1 490 bpꎻndhF 基因距离 IRb / SSC 2.4 全叶绿体基因组比对分析
边界 113 ~ 119 bpꎻ所 有 样 品 的 ycf1 基 因 均 横 跨 以黄草乌为参考ꎬ使用 mVISTA 在线工具对
SSC 区和 IRa 区ꎬ并且 ycf1 基因大部分位于 SSC 区 10 个栽培品的叶绿体基因组进行全序列比对ꎬ以
(4 012 ~ 4 027 bp)ꎬ均有1 286 bp 位于 IRa 区ꎻ在 分析 10 个栽培品的叶绿体基因组序列差异ꎮ 由
10 个样品中 IRa / LSC 边界均处于 rpl2 和 trnH 基 图 4 可知ꎬ10 个栽培品的基因组高度保守ꎬ其中ꎬ
因间ꎬtrnH 基因距离 IRa / LSC 边界 120 ~ 126 bpꎮ IR 区更为保守ꎬ编码区相较于非编码区更保守ꎮ