Page 70 - 《广西植物》2020年第2期
P. 70
2 期 王伟妍等: 滇龙胆 8 ̄羟香叶醇氧化还原酶基因的克隆与表达分析 2 0 7
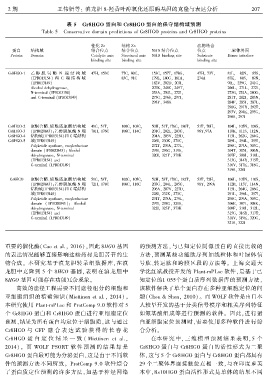
表 5 Gr8HGO 蛋白和 Cr8HGO 蛋白的保守结构域预测
Table 5 Conservative domain predictions of Gr8HGO proteins and Cr8HGO proteins
催化 Zn 结构 Zn 底物结合
蛋白 结构域 结合位点 结合位点 NAD 结合位点 位点 二聚体界面
Protein Domain Catalytic znic Structural znic NAD binding site Substrate Dimer interface
binding site binding site binding site
Gr8HGO ̄1 乙 醇 脱 氢 酶 N 端 结 构 域 47Hꎬ 153C 77Cꎬ 80Cꎬ 153Cꎬ 157Tꎬ 178Gꎬ 47Hꎬ 73Yꎬ 81Lꎬ 82Nꎬ 85Sꎬ
(IPR013154) 和 C 端 结 构 域 83Cꎬ 91C 179Lꎬ 180Gꎬ 181Aꎬ 274A 87Kꎬ 88Sꎬ 89Nꎬ
(IPR013149) 182Vꎬ 202Dꎬ 203Iꎬ 90Lꎬ 239Gꎬ 241Gꎬ
Alcohol dehydrogenaseꎬ 207Kꎬ 248Cꎬ 249Tꎬ 266Lꎬ 271Lꎬ 272Iꎬ
N ̄terminal (IPR013154) 253Aꎬ 254Lꎬ 272Iꎬ 273Gꎬ 274Aꎬ 280Gꎬ
and C ̄terminal (IPR013149) 273Gꎬ 296Sꎬ 297Iꎬ 281Tꎬ 282Iꎬ 283Nꎬ
298Yꎬ 348K 284Fꎬ 285Iꎬ 287Lꎬ
290Gꎬ 291Rꎬ 292Tꎬ
293Vꎬ 294Kꎬ 295Gꎬ
296Sꎬ 297I
Gr8HGO ̄2 聚酮合酶、烯酰还原酶结构域 49Cꎬ 51Tꎬ 100Cꎬ 103Cꎬ 50Hꎬ 51Tꎬ176Cꎬ 180Tꎬ 51Tꎬ 70Hꎬ 104Lꎬ 105Nꎬ 108Sꎬ
Gr8HGO ̄3 (IPR020843)ꎻ乙醇脱氢酶 N 端 70Hꎬ 176C 106Cꎬ 114C 201Gꎬ 202Lꎬ 203Gꎬ 96Yꎬ97A 110Kꎬ 111Sꎬ 112Nꎬ
Gr8HGO ̄4 结构域(IPR013154)和 C 端结构 204Aꎬ 205Vꎬ 225Dꎬ 113Lꎬ 262Gꎬ 264Gꎬ
Gr8HGO ̄5 域(IPR013149) 226Iꎬ 230Kꎬ 271Cꎬ 289Lꎬ 294Lꎬ 295Iꎬ
Polyketide synthaseꎬ enoylreductase 272Tꎬ 276Aꎬ 277Lꎬ 296Gꎬ 297Aꎬ 303Gꎬ
domain (IPR020843)ꎻ Alcohol 295Iꎬ 296Gꎬ 319Sꎬ 304Tꎬ 305Iꎬ 306Nꎬ
dehydrogenaseꎬ N ̄terminal 320Iꎬ 321Yꎬ 371K 307Fꎬ 308Iꎬ 310Lꎬ
(IPR013154) and 313Gꎬ 314Rꎬ 315Tꎬ
C ̄terminal (IPR013149) 316Vꎬ 317Kꎬ 318Gꎬ
319Sꎬ 320I
Cr8HGO 聚酮合酶、烯酰还原酶结构域 51Cꎬ 53Tꎬ 102Cꎬ 105Cꎬ 52Hꎬ 53Tꎬ178Cꎬ 182Tꎬ 53Tꎬ 72Hꎬ 106Lꎬ 107Nꎬ 110Sꎬ
(IPR020843)ꎻ乙醇脱氢酶 N 端 72Hꎬ 178C 108Cꎬ 116C 203Gꎬ 204Lꎬ 205Gꎬ 98Yꎬ 299A 112Rꎬ 113Tꎬ 114Nꎬ
结构域(IPR013154)和 C 端结构 206Aꎬ 207Vꎬ 227Dꎬ 115Lꎬ 264Gꎬ 266Gꎬ
域(IPR013149) 228Iꎬ 232Kꎬ 273Cꎬ 291Lꎬ 296Lꎬ 297Iꎬ
Polyketide synthaseꎬ enoylreductase 274Tꎬ 278Aꎬ 279Lꎬ 298Gꎬ 299Aꎬ 305Gꎬ
domain ( IPR020843 )ꎻ Alcohol 297Iꎬ 298Gꎬ 321Sꎬ 306Eꎬ 307Iꎬ 308Kꎬ
dehydrogenaseꎬ N ̄terminal 322Iꎬ 323Yꎬ 373K 309Fꎬ 310Iꎬ 312Lꎬ
(IPR013154) and 315Gꎬ 316Rꎬ 317Tꎬ
C ̄terminal (IPR013149) 318Vꎬ 319Kꎬ 320Gꎬ
321Sꎬ 322I
重要的催化酶(Cao et al.ꎬ 2016)ꎬ因此 8HGO 基因 的预测方法ꎬ与已知定位同源蛋白的直接比较的
的表达情况能够直接影响这些药材龙胆苦苷的生 方法ꎬ预测某些功能肽序列如线粒体和叶绿体信
物合成ꎮ 本研究基于滇龙胆转录组数据库ꎬ在滇 号肽、转运肽和跨膜区段的方法等ꎮ 上海交通大
龙胆中克隆到 5 个 8HGO 基因ꎬ表明在滇龙胆中 学沈红斌教授开发的 Plant ̄mPLoc 软件ꎬ是基于已
8HGO 基因可能存在功能冗余现象ꎮ 知定位的1 055个蛋白质序列数据库的预测方法ꎬ
高效的途径工程需要不同途径组分的细胞和 该软件解决了单个蛋白存在多种亚细胞定位的问
亚细胞组织的精确知识( Miettinen et al.ꎬ 2014)ꎮ 题(Chou & Shenꎬ 2010)ꎮ 而 WOLF 软件是由日本
本研究使用 Plant ̄mPLoc 和 ProtComp 9.0 软件对 5 人较早开发的基于分类信号模序和相关序列特征
个 Gr8HGO 蛋白和 Cr8HGO 蛋白进行亚细胞定位 如氨基酸组成等进行预测的软件ꎮ 因此ꎬ进行蛋
预测ꎬ结果为所有蛋白均定位于细胞质ꎬ这与通过 白亚细胞定位预测时ꎬ需要使用多种软件进行综
Cr8HGO 与 GFP 融 合 表 达 试 验 获 得 的 长 春 花 合分析ꎮ
Cr8HGO 蛋 白 定 位 结 果 一 致 ( Miettinen et al.ꎬ 在本研 究 中ꎬ 三 维 模 型 预 测 结 果 表 明ꎬ5 个
2014)ꎮ 而 WOLF PSORT 软 件 预 测 的 结 果 却 是 Gr8HGO 蛋白与 Cr8HGO 蛋白的活性形式为二聚
Gr8HGO 蛋白最可能为分泌蛋白ꎬ这是由于不同软 体ꎬ这与 5 个 Gr8HGO 蛋白与 Cr8HGO 蛋白都具有
件的预测方法不同所致ꎮ ProtComp 9.0 软件综合 29 个二聚体界面接触位点相一致ꎬ与在印度萝芙
了蛋白质定位预测的许多方法ꎬ如基于神经网络 木中ꎬRs10HGO 蛋白活性形式是单体的结果不同