Page 126 - 《广西植物》2023年第10期
P. 126
1 8 8 0 广 西 植 物 43 卷
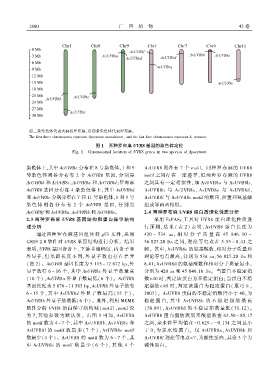
前三条染色体代表大麻状罗布麻ꎬ后四条染色体代表罗布麻ꎮ
The first three chromosomes represent Apocynum cannabinumꎬ and the last four chromosomes represent A. venetum.
图 1 两种罗布麻 UVR8 基因的染色体定位
Fig. 1 Chromosomal location of UVR8 genes in two species of Apocynum
染色体上ꎬ其中 AcUVR8e 分布在 8 号染色体ꎬ1 和 9 AcUVR8 则各有 7 个 motifꎮ 同种罗布麻的 UVR8
号染色体则各分布有 2 个 AcUVR8 基因ꎬ 分别是 motif 之间存在一定差异ꎬ但两种罗布麻的 UVR8
AcUVR8b 和 AcUVR8cꎬAcUVR8a 和 AcUVR8dꎻ罗布麻 之间具有一定相似性ꎬ如 AvUVR8a 与 AcUVR8b、
AvUVR8 基因分布在 4 条染色体上ꎬ其中 AvUVR8d AvUVR8b 与 AcUVR8a、 AvUVR8e 与 AcUVR8d、
和 AvUVR8c 分别分布在 7 和 11 号染色体ꎬ1 和 9 号 AvUVR8f 与 AcUVR8c motif 的数目、位置和氨基酸
染色 体 则 各 分 布 有 2 个 AvUVR8 基 因ꎬ 分 别 是 组成等两两相似ꎮ
AvUVR8f 和 AvUVR8a、AvUVR8b 和 AvUVR8eꎮ 2.4 两种罗布麻 UVR8 蛋白质理化性质分析
2.3 两种罗布麻 UVR8 基因结构和蛋白保守结构 采用 ExPASy 工具对 UVR8 蛋白理化性质进
域分析 行预 测ꎬ 结 果 ( 表 2) 表 明ꎬ AvUVR8 蛋 白 长 度 为
通过两种罗布麻基因组注释 gff3 文件ꎬ采用 420 ~ 534 aaꎬ 相 对 分 子 质 量 在 45 846. 10 ~
GSDS 2.0 软件对 UVR8 基因结构进行分析ꎮ 结果 56 827.20 Da 之间ꎬ理论等电点在 5.55 ~ 8.41 之
表明ꎬUVR8 基因皆含上、下游非编码区、内含子和 间ꎮ 其中ꎬAvUVR8c 的氨基酸数、相对分子质量和
外显子ꎬ但基因长度不同ꎬ外显子数也存在差异 理论等电点最高ꎬ分别为 534 aa、56 827.20 Da 和
(图 2)ꎮ AvUVR8 基因长度为 5 115 ~ 12 672 bpꎬ外 8.41ꎻAvUVR8d 的氨基酸数和相对分子质量最小ꎬ
显子数有 6 ~ 16 个ꎬ其中 AvUVR8c 外显子数最高 分别为 420 aa 和 45 846.10 Daꎮ 当蛋白不稳定指
(16 个)ꎬAvUVR8a 外显子数最低(6 个)ꎮ AcUVR8 数>40 时ꎬ判定该蛋白为不稳定蛋白ꎻ当蛋白不稳
基因长度为 5 876 ~ 11 585 bpꎬAcUVR8 外显子数有 定指数<40 时ꎬ判定该蛋白为稳定蛋白( 夏巧玉ꎬ
6 ~ 15 个ꎬ其中 AcUVR8d 外显子数最高( 15 个)ꎬ 2007)ꎮ AvUVR8 蛋白的不稳定指数皆小于 40ꎬ为
AcUVR8b 外显子数最低(6 个)ꎮ 此外ꎬ利用 MEME 稳定 蛋 白ꎬ 其 中 AvUVR8b 的 不 稳 定 指 数 最 高
软件分析 UVR8 蛋白保守结构域( motif)ꎬmotif 设 (38.89)ꎬAvUVR8d 的不稳定指数最低( 31.12)ꎮ
为 7ꎬ其他参数为默认值ꎮ 由图 3 可知ꎬAvUVR8 AvUVR8 蛋白脂肪族氨基酸指数在 63.50 ~ 85.17
的 motif 数为 4 ~ 7 个ꎬ其中 AvUVR8b、AvUVR8e 和 之间ꎬ亲水性平均值在- 0.629 ~ - 0.134 之间且小
AvUVR8f 的 motif 数 最 多 ( 7 个)ꎬ AvUVR8c motif 于 0ꎬ为 亲 水 性 蛋 白ꎮ 仅 AvUVR8a、 AvUVR8b 和
数最少(4 个)ꎮ AcUVR8 的 motif 数为 6 ~ 7 个ꎬ其 AvUVR8f 理论等电点<7ꎬ为酸性蛋白ꎬ其余 3 个为
中 AcUVR8b 的 motif 数 最 少 ( 6 个)ꎬ 其 他 4 个 碱性蛋白ꎮ